Mocapy++ 是一个用于训练和使用贝叶斯网络的机器学习工具包。它已被用于开发生物分子结构的概率模型。该项目的目的是开发一个 Mocapy++ 的 Python 接口,并将它集成到 Biopython 中。这将允许使用从数据库中提取的数据来训练概率模型。Mocapy++ 与 Biopython 的集成将为蛋白质结构预测、设计和模拟领域提供强大的支持。
发现生物分子的结构是生物学中最大的问题之一。给定一个氨基酸或碱基序列,其三维结构是什么?一种生物分子结构预测方法是构建概率模型。贝叶斯网络是一种概率模型,它由一组变量及其联合概率分布组成,以有向无环图的形式表示。动态贝叶斯网络是一种表示变量序列的贝叶斯网络。这些序列可以是时间序列或符号序列,例如蛋白质序列。方向统计主要关注平面或三维空间中的单位向量观测。样本空间通常是圆或球体。必须有特殊的定向方法来考虑样本空间的结构。图形模型和方向统计的结合使得可以开发生物分子结构的概率模型。通过使用具有方向输出的动态贝叶斯网络,可以构建序列和结构的联合概率分布。生物分子结构可以在几何上自然、连续的空间中表示。Mocapy++ 是一个开源工具包,用于使用支持方向统计的动态贝叶斯网络进行推理和学习。Mocapy++ 非常适合构建生物分子结构的概率模型;它已被用于开发原子级蛋白质和 RNA 结构模型。Mocapy++ 被用于几个高影响力的出版物,并将成为即将发布的分子建模包 Phaistos 的核心。该项目的目的是开发一个非常有用的 Mocapy++ 的 Python 接口,并将该接口集成到 Biopython 项目中。通过 Bio.PDB 模块,Biopython 提供了出色的数据挖掘生物分子结构数据库的功能。集成 Mocapy++ 和 Biopython 将允许使用从数据库中提取的数据来训练概率模型。将 Mocapy++ 集成到 Biopython 将为研究人员创建一个强大的工具包,让他们能够快速实施和测试新想法、尝试各种方法并改进其方法。它将为生物分子结构预测、设计和模拟领域提供强大的支持。
Michele Silva (michele.silva@gmail.com)
指导老师
Thomas Hamelryck
Eric Talevich
’'’了解 SEM 和方向统计 ‘’’
’'’研究 Mocapy++ 的用例 ‘’’
’'’使用 Mocapy++ ‘’’
’'’设计 Mocapy++ 的 Python 接口 ‘’’
’'’实现 Python 绑定 ‘’’
’'’探索 Mocapy++ 的应用 ‘’’
托管在 gSoC11 Mocapy 分支
已经有人努力使用 Swig 为 Mocapy++ 提供绑定。但是,如果需要性能,Swig 不是最佳选择。Sage 项目旨在为 Mathematica 或 Maple 提供一个开源替代方案。Cython 是与 Sage 共同开发的(虽然它是一个独立的项目),因此它是基于 Sage 的需求的。他们尝试了 Swig,但由于性能问题而放弃了它。根据 Sage 编程指南,“最初的想法是用 C++ 为 SAGE 编写需要快速运行的代码,然后用 SWIG 包装它。这最终停止了,因为结果不够快。首先,用 C++ 编写代码本身就存在开销。其次,SWIG 在 Python 和实际执行工作的代码之间生成几层代码”。这是在 2004 年写的,但似乎情况没有太大变化。我考虑 Swig 的唯一原因是为了将来在 BioJava 和 BioRuby 项目中包含 Mocapy++ 绑定。
Boost Python 非常全面,并且被 Python 社区广泛认可。我会因为它被广泛使用和测试而选择它。我会因为调试困难和构建系统复杂而拒绝它。我认为仅仅为了创建 Python 绑定而添加一个 Boost 依赖项并不值得,但是由于 Mocapy++ 已经依赖于 Boost,使用它就成为一个更有吸引力的选择。根据我个人的经验,Boost Python 非常成熟,并且在使用它时没有任何限制。在性能方面,Cython 仍然优于它。看看 Cython C++ 包装基准测试 并检查 Cython 与 Boost Python 的计时。还有一些 基准测试比较了 Swig 和 Boost Python。
根据网络上提供的几个基准测试,它比其他创建 C++ 代码 Python 绑定的选项快得多。检查 Cython 和 Boost.Python 之间的简单基准测试。它也非常干净、简单,但功能强大。Python 关于移植扩展模块的文档中提到了 cython:“如果您正在编写一个新的扩展模块,您可能需要考虑使用 Cython。”Cython 现在支持与 numpy 数组的有效交互。它是一种年轻但正在发展的语言,我一定会尝试它,因为它轻量级且速度快。
由于 Boost 受到了良好的支持,并且 Mocapy++ 已经依赖于它,因此我们决定使用 Boost.Python 来进行绑定。
有关更多信息,请参阅 Mocapy++Biopython - 想法集。
原型的源代码在 gSoC11 分支上: http://mocapy.svn.sourceforge.net/viewvc/mocapy/branches/gSoC11/bindings_prototype/
为一些 Mocapy++ 功能提供绑定以及几个示例,以查找可能的实现和性能问题。
过程
Mocapy++ 的接口保持不变,因此测试看起来与 Mocapy/examples 中的测试类似。
在原型中,所有绑定都在单个模块中实现。对于实际实现,我们可以镜像 src 包的结构,为每个包(如 discrete、inference 等)提供独立的绑定。
能够实现运行示例所需的所有功能。在为 MDArray 的向量创建绑定时,无法使用 vector_indexing_suite。必须实现一些运算符(在 MDArray 中),以便将可索引的 C++ 容器导出到 Python。
在 Python 中实现了两个使用离散节点的 Mocapy++ 示例。公开 Mocapy 的数据结构和算法没有问题。Python 版本的性能与原始 Mocapy++ 非常接近。
有关更多详细信息,请查看 Mocapy++ 绑定原型 报告。
’’’ 数据结构 ‘’’
Mocapy 使用一个内部数据结构来表示数组:MDArray。为了使用户更轻松地与 Mocapy 的 API 交互,决定提供一个接受 numpy 数组的接口。因此,需要实现 numpy 数组和 MDArray 之间的转换。
通过使用 Boost.Python to_python_converter 完成了从 MDArray 到 python 的转换。我们实现了一个模板方法 convert_MDArray_to_numpy_array,它将任何基本类型的 MDArray 转换为相应的 numpy 数组。为了执行转换,将原始数组的形状和内部数据复制到一个新的 numpy 数组中。
使用 Numpy 数组 API 创建了 numpy 数组。使用现有数据 (PyArray_SimpleNewFromData) 创建新的 PyArrayObject 不会复制数组数据,它只会存储指向它的指针。因此,只有在没有对该对象的引用时才能释放数据。这是通过使用 Capsule 完成的。除了封装数据外,胶囊还存储一个在数组被销毁时使用的析构函数。PyArrayObject 有一个名为“base”的字段,它指向胶囊。
从 Python 到 C++ 的转换,即从 numpy 数组创建 MDArray 稍微复杂一些。Boost.Python 将提供一块内存,新创建的 C++ 对象必须在其中就地构造。有关更多详细信息,请参阅 如何编写 boost.python 转换器 文章。
还实现了基本类型 (double、int…) 的 std::vector 和 Python 列表之间的转换。对于 std::vector 的自定义类型,例如 Node,未执行到 Python 列表的转换。如果以与基本类型相同的方式完成,则会引发类型错误:“TypeError: No to_python (by-value) converter found for C++ type”。当使用 vector_indexing_suite 时,这个问题已经解决。参见 包装 std::vector<AbstractClass*>。使用 vector_indexing_suite 的唯一不便之处是创建新的类型(如 vector_Node),而不是使用标准的 Python 列表。
转换的代码位于 mocapy_data_structures 模块中。
’’’ 核心功能 ‘’’
mocapy Python 包遵循 Mocapy 当前的源代码树。 每个包都创建了一个包含绑定的共享库。 这样可以加快编译速度并简化调试。 此外,如果只创建了一个库,则无法定义包。
每个库都称为 libmocapy_
绑定代码可以在 绑定目录 中找到。
目前,正在开发对新创建接口的测试。 在框架包下已经实现了一些测试:mocapy/framework/tests
’’’ 数据结构 ‘’’
在实现对剩余 Mocapy++ 功能的绑定时,使用指向 mdarray 的指针和引用的方法出现了问题
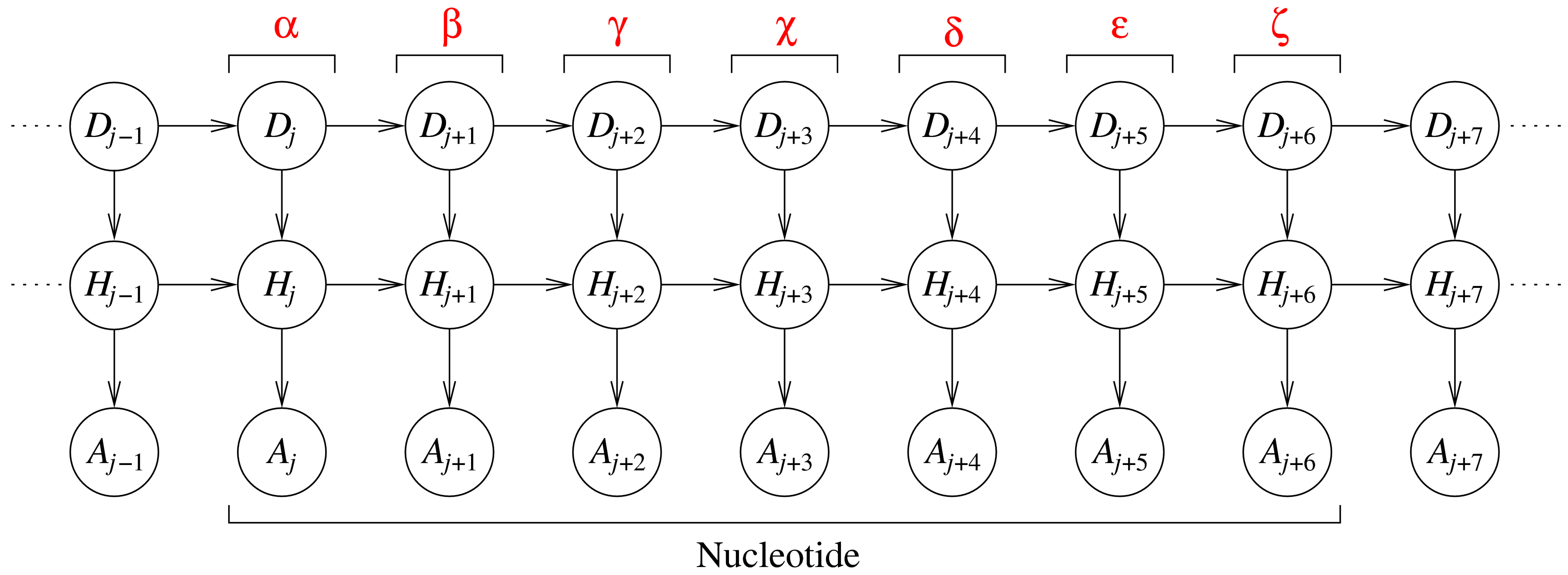 DBN 代表了九个连续的二面角,其中七个中心角来自单个核苷酸。每个切片 j(三变量的一列)对应于 RNA 片段中的一个二面角。每个切片中的变量是:角度标识符 Dj、隐藏变量 Hj 和角度变量 Aj。角度标识符跟踪切片所代表的二面角,而角度节点对实际的二面角值进行建模。隐藏节点会在整个序列中所有角度之间产生依赖关系(而不仅仅是在连续切片中的角度之间)。Barnacle 的原始源代码(包含一个用 Python 编写的嵌入式 Mocapy 版本)可以在 <http://sourceforge.net/projects/barnacle-rna> 中找到。Barnacle 的修改版本(修改为与 Mocapy 绑定一起工作)可以在 <https://github.com/mchelem/biopython/tree/master/Bio/PDB/Barnacle> 中找到。这是一个使用示例: ``` python model = Barnacle("ACCU") model.sample() print("log likelihood = ", model.get_log_likelihood()) model.save_structure("structure01.pdb") ``` #### TorusDBN Wouter Boomsma, Kanti V. Mardia, Charles C. Taylor, Jesper Ferkinghoff-Borg, Anders Krogh, and Thomas Hamelryck. 局部蛋白质结构的生成性概率模型。Proc Natl Acad Sci U S A. 2008 年 7 月 1 日;105(26): 8932–8937。<http://www.ncbi.nlm.nih.gov/pmc/articles/PMC2440424/> TorusDBN 的目标是根据生物分子的氨基酸序列预测其 3D 结构。它是一个关于蛋白质在原子细节上局部序列-结构偏好的连续概率模型。蛋白质的主链可以用一系列二面角对 φ 和 ψ 来表示,这些二面角在 [Ramachandran 图](http://en.wikipedia.org/wiki/Ramachandran_plot) 中是众所周知的。两个角度(都取值范围从 -180° 到 180°)定义了环面上的一个点。因此,蛋白质的主链结构可以完全参数化为一系列这样的点。
DBN 代表了九个连续的二面角,其中七个中心角来自单个核苷酸。每个切片 j(三变量的一列)对应于 RNA 片段中的一个二面角。每个切片中的变量是:角度标识符 Dj、隐藏变量 Hj 和角度变量 Aj。角度标识符跟踪切片所代表的二面角,而角度节点对实际的二面角值进行建模。隐藏节点会在整个序列中所有角度之间产生依赖关系(而不仅仅是在连续切片中的角度之间)。Barnacle 的原始源代码(包含一个用 Python 编写的嵌入式 Mocapy 版本)可以在 <http://sourceforge.net/projects/barnacle-rna> 中找到。Barnacle 的修改版本(修改为与 Mocapy 绑定一起工作)可以在 <https://github.com/mchelem/biopython/tree/master/Bio/PDB/Barnacle> 中找到。这是一个使用示例: ``` python model = Barnacle("ACCU") model.sample() print("log likelihood = ", model.get_log_likelihood()) model.save_structure("structure01.pdb") ``` #### TorusDBN Wouter Boomsma, Kanti V. Mardia, Charles C. Taylor, Jesper Ferkinghoff-Borg, Anders Krogh, and Thomas Hamelryck. 局部蛋白质结构的生成性概率模型。Proc Natl Acad Sci U S A. 2008 年 7 月 1 日;105(26): 8932–8937。<http://www.ncbi.nlm.nih.gov/pmc/articles/PMC2440424/> TorusDBN 的目标是根据生物分子的氨基酸序列预测其 3D 结构。它是一个关于蛋白质在原子细节上局部序列-结构偏好的连续概率模型。蛋白质的主链可以用一系列二面角对 φ 和 ψ 来表示,这些二面角在 [Ramachandran 图](http://en.wikipedia.org/wiki/Ramachandran_plot) 中是众所周知的。两个角度(都取值范围从 -180° 到 180°)定义了环面上的一个点。因此,蛋白质的主链结构可以完全参数化为一系列这样的点。  圆形节点表示随机变量。箭头沿着的矩形框说明了它们之间条件概率分布的性质。一个隐藏节点会发出角度对、氨基酸信息、二级结构标签和顺式/反式信息。TorusDBN 模型最初是作为 backboneDBN 包的一部分实现的,该包可以在 <http://sourceforge.net/projects/phaistos/> 中免费获得。在本项目中实现了一个新的 TorusDBN 模型版本,可以在 <https://github.com/mchelem/biopython/tree/master/Bio/PDB/TorusDBN> 中找到。TorusDBNTrainer 可用于使用给定的训练集训练模型: ``` python trainer = TorusDBNTrainer() trainer.train(training_set) # training_set 是一个文件列表 model = trainer.get_model() ``` 然后可以使用该模型来采样新序列: ``` python model.set_aa("ACDEFGHIK") model.sample() print(model.get_angles()) # 采样的角度。 ``` 创建模型时,可以创建一个指定隐藏节点大小的新 DBN,或者从文件中加载 DBN。 ``` python model = TorusDBNModel() model.create_dbn(hidden_node_size=10) model.save_dbn("test.dbn") ``` ``` python model = TorusDBNModel() model.load_dbn("test.dbn") model.set_aa("ACDEFGHIK") model.sample() print(model.get_angles()) # 采样的角度。 ``` 还可以使用 find_optimal_model 方法选择隐藏节点的最佳大小: ``` python trainer = TorusDBNTrainer() hidden_node_size, IC = trainer.find_optimal_model(training_set) model = trainer.get_model() ``` IC 是 [贝叶斯信息准则](http://en.wikipedia.org/wiki/Bayesian_information_criterion) (BIC) 或 [赤池信息准则](http://en.wikipedia.org/wiki/Akaike_information_criterion) (AIC)(默认值为 BIC。AIC 可以通过设置 use_aic 标志来指定)。有关模型 API 的更多详细信息,请参阅测试文件:<https://github.com/mchelem/biopython/blob/master/Tests/test_TorusDBNTrainer.py> 和 <https://github.com/mchelem/biopython/blob/master/Tests/test_TorusDBNModel.py>。 ### 性能 对在 C++ 和 Python 中实现的测试用例进行了性能测量。测试在一台具有以下规格的计算机上运行:酷睿 2 双核 T7250 2.00GHz、内存双通道 4.0GB(2x2048)667 MHz DDR2 SDRAM、硬盘 200GB 7200RPM。没有明显的性能差异。对于两种实现,消耗大部分 CPU 时间的方法都是相同的:
圆形节点表示随机变量。箭头沿着的矩形框说明了它们之间条件概率分布的性质。一个隐藏节点会发出角度对、氨基酸信息、二级结构标签和顺式/反式信息。TorusDBN 模型最初是作为 backboneDBN 包的一部分实现的,该包可以在 <http://sourceforge.net/projects/phaistos/> 中免费获得。在本项目中实现了一个新的 TorusDBN 模型版本,可以在 <https://github.com/mchelem/biopython/tree/master/Bio/PDB/TorusDBN> 中找到。TorusDBNTrainer 可用于使用给定的训练集训练模型: ``` python trainer = TorusDBNTrainer() trainer.train(training_set) # training_set 是一个文件列表 model = trainer.get_model() ``` 然后可以使用该模型来采样新序列: ``` python model.set_aa("ACDEFGHIK") model.sample() print(model.get_angles()) # 采样的角度。 ``` 创建模型时,可以创建一个指定隐藏节点大小的新 DBN,或者从文件中加载 DBN。 ``` python model = TorusDBNModel() model.create_dbn(hidden_node_size=10) model.save_dbn("test.dbn") ``` ``` python model = TorusDBNModel() model.load_dbn("test.dbn") model.set_aa("ACDEFGHIK") model.sample() print(model.get_angles()) # 采样的角度。 ``` 还可以使用 find_optimal_model 方法选择隐藏节点的最佳大小: ``` python trainer = TorusDBNTrainer() hidden_node_size, IC = trainer.find_optimal_model(training_set) model = trainer.get_model() ``` IC 是 [贝叶斯信息准则](http://en.wikipedia.org/wiki/Bayesian_information_criterion) (BIC) 或 [赤池信息准则](http://en.wikipedia.org/wiki/Akaike_information_criterion) (AIC)(默认值为 BIC。AIC 可以通过设置 use_aic 标志来指定)。有关模型 API 的更多详细信息,请参阅测试文件:<https://github.com/mchelem/biopython/blob/master/Tests/test_TorusDBNTrainer.py> 和 <https://github.com/mchelem/biopython/blob/master/Tests/test_TorusDBNModel.py>。 ### 性能 对在 C++ 和 Python 中实现的测试用例进行了性能测量。测试在一台具有以下规格的计算机上运行:酷睿 2 双核 T7250 2.00GHz、内存双通道 4.0GB(2x2048)667 MHz DDR2 SDRAM、硬盘 200GB 7200RPM。没有明显的性能差异。对于两种实现,消耗大部分 CPU 时间的方法都是相同的: 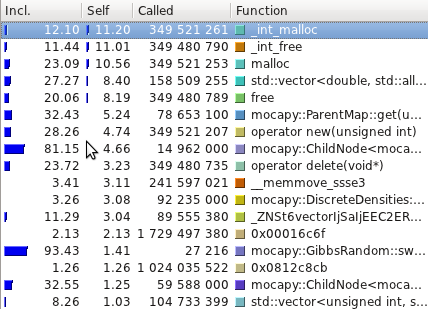
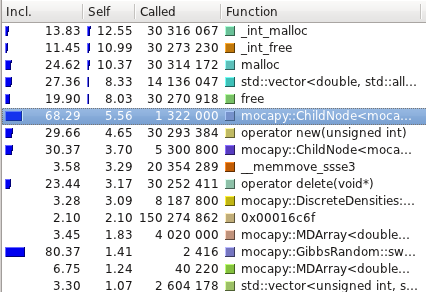 性能测试使用 [Callgrind](http://valgrind.org/info/tools.html#callgrind) 进行,并使用 [Kcachegrind](http://kcachegrind.sourceforge.net/) 可视化。以下是 Mocapy 提供的示例的平均运行时间(10 次运行): | 测试名称 | C++ (s) | Python (s) | |----------------------------|---------|------------| | hmm_simple | 0.52 | 0.58 | | hmm_discrete | 48.12 | 43.45 | | discrete_hmm_with_prior | 55.95 | 50.09 | | hmm_dirichlet | 340.72 | 353.98 | | hmm_factorial | 0.01 | 0.12 | | hmm_gauss_1d | 53.97 | 63.39 | | hmm_gauss | 16.02 | 16.96 | | hmm_multinomial | 134.64 | 125.83 | | hmm_poisson | 11.00 | 10.60 | | hmm_vonmises | 7.22 | 7.36 | | hmm_torus | 53.79 | 53.65 | | hmm_kent | 61.35 | 61.06 | | hmm_bippo | 40.66 | 41.81 | | infenginehmm | 0.01 | 0.12 | | infenginemm | 0.01 | 0.15 | #### TorusDBN 尽管 PDB 文件以 mocapy 可以理解的格式读取、解析和转换,但最耗时的方法是那些在采样过程中执行数学运算的方法(例如 Chebyshev 和 exp)。
性能测试使用 [Callgrind](http://valgrind.org/info/tools.html#callgrind) 进行,并使用 [Kcachegrind](http://kcachegrind.sourceforge.net/) 可视化。以下是 Mocapy 提供的示例的平均运行时间(10 次运行): | 测试名称 | C++ (s) | Python (s) | |----------------------------|---------|------------| | hmm_simple | 0.52 | 0.58 | | hmm_discrete | 48.12 | 43.45 | | discrete_hmm_with_prior | 55.95 | 50.09 | | hmm_dirichlet | 340.72 | 353.98 | | hmm_factorial | 0.01 | 0.12 | | hmm_gauss_1d | 53.97 | 63.39 | | hmm_gauss | 16.02 | 16.96 | | hmm_multinomial | 134.64 | 125.83 | | hmm_poisson | 11.00 | 10.60 | | hmm_vonmises | 7.22 | 7.36 | | hmm_torus | 53.79 | 53.65 | | hmm_kent | 61.35 | 61.06 | | hmm_bippo | 40.66 | 41.81 | | infenginehmm | 0.01 | 0.12 | | infenginemm | 0.01 | 0.15 | #### TorusDBN 尽管 PDB 文件以 mocapy 可以理解的格式读取、解析和转换,但最耗时的方法是那些在采样过程中执行数学运算的方法(例如 Chebyshev 和 exp)。 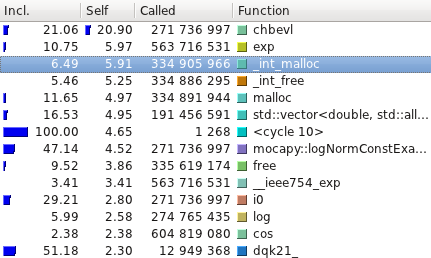 该模型已使用一个训练集进行训练,该训练集包含大约 950 个链,最大同源性为 20%,分辨率低于 1.6 Å,R 因子低于 25%。读取和训练整个数据集大约需要 67 分钟。生成的 DBN 可在 <https://github.com/mchelem/biopython/blob/master/Tests/TorusDBN/pisces_dataset.dbn> 中获得,可以像上面 TorusDBN 部分所述的那样直接加载到模型中。 ### 未来工作 虽然夏天已经结束,但工作仍在继续……我还有很多事情要做: - 测试训练后的模型以检查它们在蛋白质结构预测中的有效性。- 尝试减少动态分配,因为它会导致很多运行时间。- 保证绑定中没有内存泄漏。
该模型已使用一个训练集进行训练,该训练集包含大约 950 个链,最大同源性为 20%,分辨率低于 1.6 Å,R 因子低于 25%。读取和训练整个数据集大约需要 67 分钟。生成的 DBN 可在 <https://github.com/mchelem/biopython/blob/master/Tests/TorusDBN/pisces_dataset.dbn> 中获得,可以像上面 TorusDBN 部分所述的那样直接加载到模型中。 ### 未来工作 虽然夏天已经结束,但工作仍在继续……我还有很多事情要做: - 测试训练后的模型以检查它们在蛋白质结构预测中的有效性。- 尝试减少动态分配,因为它会导致很多运行时间。- 保证绑定中没有内存泄漏。